《生物信息学》复习提纲
《生物信息学》主要知识点
一、基本名词和概念
1、bioinformatics 生物信息学,狭义的生物信息学是指将计算机科学和数学应用于生物大分子信息的获取、加工、存储、分类、检索与分析,以达到理解这些生物大分子信息的生物学意义的一门交叉学科。广义上的生物信息学是指运用计算机技术,处理、分析生物学数据,以揭示生物学数据背后蕴藏的意义的所有知识体系。
2、ORF Open Reading Frame,开放阅读框,是指在给定的阅读框架中,不包含终止密码子的一串DNA序列
3、CDS Coding sequence,基因的编码区(也叫Coding region),是指DNA或RNA中由外显子组成,编码蛋白质的部分。
4、UTR Untranslated Regions,即非翻译区,是指mRNA分子两端的非编码片段,包括5'-UTR(或称“前导序列”)和3'-UTR(或称“尾随序列”)

5、genome 基因组,是指包含在一种生物的单倍体细胞中的全套染色体DNA(部分病毒是RNA)中的全部遗传信息,包括基因和非编码DNA。
6、proteomics 蛋白质组学,对特定的通路、细胞器、细胞、组织、器官和肌体中包含的所有蛋白质,进行鉴定、表征和定量,提供关于该系统准确和全面数据的学科。
7、transcriptome 转录组,也称为“转录物组”,广义上指在相同环境(或生理条件)下的一个细胞、组织或生物体中出现的所有RNA的总和,包括mRNA、rRNA、tRNA及非编码RNA;狭义上则指细胞所能转录出的所有mRNA。
8、metabonomics 代谢组学,属于系统生物学的一个重要组成部分,效仿基因组学和蛋白质组学的研究思想,对生物体内所有代谢物进行定量分析,从而研究生命体对外界刺激、病理生理变化、以及本身基因突变而产生的其体内代谢物水平的多元动态反应。其研究对象大都是相对分子质量1000以内的小分子物质。
9、functional genomics 功能基因组学,是一门利用结构基因组学研究所得到的各种信息,建立和发展各种技术和实验模型来测定基因和基因组非编码序列的生物学功能的学科。
10、genomic mapping 基因组作图,就是确定界标或基因在构成基因组的每条染色体上的位置,以及同条染色体上各个界标或基因之间的相对距离。
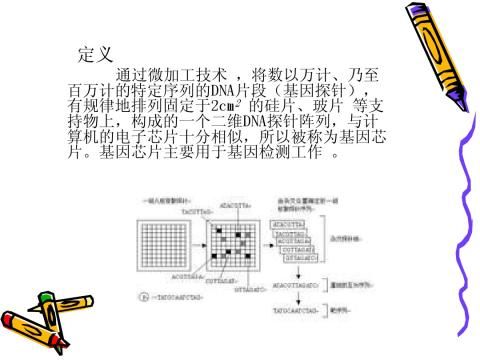
11、microarray DNA微阵列,又称基因芯片(gene chip),是由大量DNA或寡核苷酸探针密集排列所形成的探针阵列,其工作的基本原理是通过杂交检测信息。
12、nucleotide 核苷酸,是指核苷( Nucleoside)和磷酸( Phosphate groups)结合的化学物质,包括单核苷酸(如AMP、CMP等)、寡核苷酸(ADP、ATP、GTP等)和多核苷酸(DNA、RNA等)。
13、Linux 是一种自由和开放源代码的类UNIX操作系统。
14、Perl Practical Extraction and Report Language,实用报表提取语言,是一种特别擅长处理字符串文本数据的计算机编程语言,兼具脚本语言和高级语言的特征。
15、alignment 序列比对,或叫联配,是指在两条或多条序列中寻找按照相同次序排布的一连串单字符或字符模块的过程
16、BLAST basic local alignmeng search tools 同源序列比对工具的一个集合,也是一种两两序列比对算法的名称
17、phylogeny 系统发生(或系统发育),是指生物形成或进化的历史
18、Orthologs 直系同源 指来自于不同物种的由垂直家系(物种形成)进化而来的基因或蛋白,并且典型的保留与原始基因或蛋白有相同的功能。
19、Paralogs 旁系同源,是指是指同一基因组(或同系物种的基因组)中,由于始祖基因的加倍而横向(horizontal)产生的几个同源基因。
20、CADD Computer Aided Drug Design 计算机辅助药物设计
21、HMM Hidden Markov model,隐马尔科夫模型,一种用来描述含有隐含未知参数的马尔可夫过程的统计模型。
22、CpG岛 是指哺乳类生物基因组中长度为0.5~4kb的一段富含胞嘧啶(C)、鸟嘌呤(G)及使两者相连的磷酸酯键(p)成分的DNA序列,几乎都位于基因的启动子区。
二、常用生物信息学软件或在线工具
1. Clustal(或Clustal X) 多序列比对软件(X为视窗版,W为命令行版)
2. Phylip 一种命令行格式的分子系统发育分析软件,包含多种算法
3. BioEdit 一种以序列编辑与分析为主的功能比较全面的综合性软件
4. Mega 一种视窗版的序列统计和进化分析的工具包(具备web序列数据库检索和多序列比对功能)
5. Treeview 进化树图形编辑软件
6. RASMOL 三维分子结构显示和分析软件
7. Primer Premier PCR引物设计软件
8. RNAstructure 建立在Turner热力学数据基础上的RNA二级结构预测软件
9. PromoterScan 一个预测分析启动子区域的在线工具
10. CpGPlot 预测CpG岛的在线平台
11. TMHMM 一个在线分析蛋白质跨膜区的工具
12、PSIPED 采用双层反馈神经网络通过对PST-BLAST搜索同源序列来预测蛋白质二级结构的在线工具。
三、常用生物信息学数据库平台及其支撑机构
1、NCBI National Center of Biotechnology Information,美国国立生物技术信息中心,其下建立的GenBank是世界三大DNA数据库之一。
2、EBI European Bioinformatics Institute 欧洲生物信息研究所,其下的EMBL(European Molecular Biology Laboratory)数据库是世界三大DNA数据库之一。
3、DDBJ DNA Data Bank of Japan日本DNA数据库
4、AceDB 最初是为秀丽新小杆线虫建立的基因组数据库,现已发展成为一个灵活和通用的数据库管理系统,可用于包括从细菌、真菌、寄生虫、植物、昆虫、动物到人类的基因组数据库的数据分析。
5、PDB Protein Data Bank,是一个专门收录蛋白质及核酸等大分子三维结构资料的数据库。
6、KEGG Kyoto Encyclopedia of Genes and Genomes京都基因与基因组百科全书,是一个以基因与分子网络为特色的一个数据库,帮助研究者了解生物系统(如细胞,生物和生态系统)的高层次功能,优势在于它所具有的PATHWAY,将各种生化反应以网络图的形式展现。
7、ExPASy Expert Protein Analysis System,蛋白质分析专家系统,是由瑞士生物信息学研究所(Swiss Institute of Bioinformatics )维护的一个提供从序列到结构以及二维电泳等全套蛋白质组学相关操作的综合性在线服务平台。
8、CDD The Conserved Domain Database ,NCBI下的蛋白质保守结构域数据库
四、常用分子系统发育分析算法及其工具
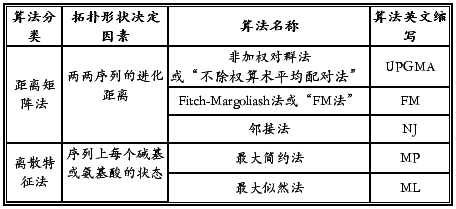
 分子进化分析软件包Phylip中的核酸序列分析程序
分子进化分析软件包Phylip中的核酸序列分析程序
 分子进化分析软件包Phylip中的蛋白质序列分析工具
分子进化分析软件包Phylip中的蛋白质序列分析工具
 分子进化分析软件Phylip中的距离矩阵计算工具neighbor
分子进化分析软件Phylip中的距离矩阵计算工具neighbor
 统计分析(Seqboot,即拨靴法或自举法,用以产生大量的数据组)
统计分析(Seqboot,即拨靴法或自举法,用以产生大量的数据组)
 分子进化分析软件包Phylip中的进化树绘制工具
分子进化分析软件包Phylip中的进化树绘制工具
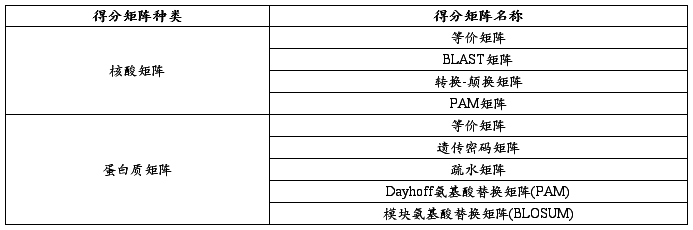
五、常用序列比对得分矩阵的种类和名称
六、基本理论和方法
(1)生物信息学研究的基本方法和前沿技术
基本方法:建立生物数据库,数据库检索,序列分析,统计模型,算法
前沿技术:数据管理技术,数据仓库、数据挖掘与数据库中的知识发现技术,图像处理与可视化技术
(2)识别基因的主要方法
1、ORF识别法 根据终止密码子出现频率、最长ORF法等辨别编码区(主要适用与原核序列)
(下述方法可用于真核序列)
2、基于密码子出现频率或密码子第三位的偏好性的预测方法
3、同源性方法
4、神经网络方法
5、隐马尔科夫模型法
6、模式判断分析法
7、动态规划方法
8、基于剪切比对的识别方法
(3)蛋白质组学与基因组学的异同点
不同点:
a、与基因组的均一性相比,蛋白质组具有多样性。即在同一生物个体的所有体细胞中基因是一样的,但在生命发育不同阶段的细胞中蛋白质种类及数量却是大相径庭,不同组织中细胞表达的蛋白质也有很大差异;
b、基因组非常稳定,而蛋白质组则是动态变化的。即同一细胞在不同时期、不同条件下,其蛋白质组也是在不断地改变之中;
c、蛋白质研究技术远比基因技术复杂和困难。
相同点:都运用组学的研究方法,强调全面性和整体性。生物信息学在其中都起到越来越重要的作用。
(4)比较基因组学的基本原理、意义和主要研究方法
基本原理:通过模式生物基因组之间或者模式生物和人类基因组之间的比较和鉴别,在一种生物基因组中找到与另一种生物某个基因功能相似的基因,从而发现新基因。
意义:为研究生物进化、分离人类遗传病的候选基因以及预测新的基因功能提供依据。
主要研究方法:系统发育概形法,Rosetta Stone法,基因邻居法
(5)蛋白质二级结构和高级结构预测方法
1、蛋白质二级结构预测方法主要有3类:
a. 结合人工神经网络、遗传算法等机器学习方法,统计氨基酸出现频率,如Chous-Fasman方法,b. 基于单一序列或多序列比对信息分析,如GOR方法和PHD方法
c. 以已知二级结构为模板,建立保守片段或位置特异性计分矩阵,通过打分预测,如PSI-BLASTP方法
2、蛋白质高级结构预测方法主要有:比较建模法(同源建模)、threading法(逆折叠法,也叫穿针引线法)、从头预测(ab initio)
(6)生物信息学在蛋白组研究中的应用有哪些方面内容?
a.编码的DNA序列的寻找与分析(分析研究对象);
b.蛋白质序列信息的获取(搜索与测序);
c.蛋白质鉴定和性质预测;
d.蛋白质序列分析;
e.蛋白质结构和功能预测;
f.数据的分析与整合:大范围基因表达分析;蛋白-蛋白相互作用;蛋白在细胞内的定位;构建通路和细胞系统;预测和发现新的知识。
材料分析
进化树手工计算和绘制
第二篇:什么是生物信息学






-
生物信息学小结
1.什么是(基因)生物信息学?目前一般意义的生物信息学是基因层次的它是一个包含着基因组信息的获取、处理、存储、分配、分析和解释的所…
-
我眼中的生物信息学
我眼中的生物信息学学院:外国语学院年级:10级班级:商务一班姓名:学号:一、生物信息学的概念从广义上来说,生物信息学从事对基因组研…
-
生物信息学专业实习总结范文
《浙江大学优秀实习总结汇编》生物信息学岗位工作实习期总结转眼之间,两个月的实习期即将结束,回顾这两个月的实习工作,感触很深,收获颇…
-
高级生物统计学学习心得
高级生物统计学课程学习总结摘要经过一学期对生物统计学的学习我对生物统计学有了进一步的理解本文主要讲述了本学期学习生物统计之后我对生…
-
生物信息学实习一
实习一序列查询实验目的1了解三大生物信息中心的资源2学会用Entre系统查找目标序列3学会用SRS系统查找目标序列实验内容一三大生…
-
生物信息学专业实习总结范文
《浙江大学优秀实习总结汇编》生物信息学岗位工作实习期总结转眼之间,两个月的实习期即将结束,回顾这两个月的实习工作,感触很深,收获颇…
-
生物信息学小结
1.什么是(基因)生物信息学?目前一般意义的生物信息学是基因层次的它是一个包含着基因组信息的获取、处理、存储、分配、分析和解释的所…
-
高级生物统计学学习心得
高级生物统计学课程学习总结摘要经过一学期对生物统计学的学习我对生物统计学有了进一步的理解本文主要讲述了本学期学习生物统计之后我对生…
-
生物信息学实习一
实习一序列查询实验目的1了解三大生物信息中心的资源2学会用Entre系统查找目标序列3学会用SRS系统查找目标序列实验内容一三大生…
-
我眼中的生物信息学
我眼中的生物信息学学院:外国语学院年级:10级班级:商务一班姓名:学号:一、生物信息学的概念从广义上来说,生物信息学从事对基因组研…